Cómo mantener a raya el patógeno vegetal Ralstonia solanacearum

Investigadores del Centro de Investigación en Agrigenómica y de la Universidad Estatal de Carolina del Norte descubren cuatro cuellos de botella que limitan la expansión de la bacteria en las plantas de tomate resistentes a la marchitez bacteriana
15.06.2020 – PUBLICACIONES
Ralstonia solanacearum es una bacteria del suelo con efectos devastadores para muchas especies de solanáceas, como por ejemplo la patata o el tomate. Actualmente, sólo hay variedades comerciales resistentes a esta bacteria de tomate, pimiento y berenjena, lo que preocupa a agricultores de todo el planeta.
Utilizando técnicas de monitoreo de la infección en vivo, investigadores del Centro de Investigación en Agrigenómica (CRAG, Barcelona) y la Universidad Estatal de Carolina del Norte (EE.UU.) han descubierto recientemente que las variedades resistentes de tomate son capaces de frenar la expansión de la bacteria en cuatro fases espaciotemporales diferentes: en la raíz, en el transporte vertical, en la difusión entre el vasos y, sorprendentemente, en la colonización de los tejidos cercanos al sistema vascular de la planta.
“Ralstonia solanacearum y otros patógenos vasculares representan un problema muy grande porqué los esfuerzos hechos hasta ahora para descubrir los genes que determinan la resistencia de las plantas a estos patógenos no han dado resultados trasladables al campo, e indican que la resistencia sería un carácter complejo, determinado por la interacción de diferentes genes. Hacía falta pues un paso atrás y entender cómo la bacteria se comporta en las plantas resistentes y en las susceptibles”, explica Nuria Sánchez Coll, jefa del grupo de investigación del CRAG de Enfermedades bacterianas de las plantas y muerte celular y una de las autoras senior del artículo publicado recientemente en Journal of Experimental Botany.
“Se puede comparar con la investigación necesaria para tratar una nueva enfermedad humana, por ejemplo, en la Covid-19 se está viendo que el virus infecta los pulmones, pero también riñones, y otras células, y que hay personas más susceptibles que otras… Con Ralstonia también nos hacía falta entender cómo se comporta el patógeno dentro de la planta, como se transporta de un órgano a otro, etc.“, añade el investigador de la Universidad de Barcelona en el CRAG Marc Valls, quien co-dirige con Sánchez Coll el grupo de investigación y el estudio.
Este trabajo establece las bases para entender los mecanismos moleculares que limitan la colonización por parte del patógeno, y que pueden proveer nuevas herramientas de precisión para hacer frente a la marchitez bacteriana.
Ralstonia solanacearum: una bacteria de cuarentena
Globalmente, R. solanacearum se considera uno de los patógenos vegetales más importantes por el gran número de especies que afecta, por su amplia distribución geográfica y por la persistencia en los terrenos infectados. La enfermedad que causa es endémica en las áreas tropicales y subtropicales.
En la Unión Europea −donde se han reportado algunos brotes de una cepa que se desarrolla también en climas fríos− es clasificado como plaga de cuarentena, por lo que está sometido a una vigilancia estricta. En España se detectó por primera vez en 1996 y desde entonces se han ido reportando casos que se han podido controlar.
La infección por R. solanacearum produce la marchitez bacteriana (podredumbre parda cuando se refiere a la infección en la patata). El patógeno, presente en el suelo, entra en la planta por las raíces y coloniza los vasos que transportan el agua y las sales minerales en las partes aéreas de la planta, haciendo un tapón que causa un marchitamiento rápido de las hojas y, finalmente, la muerte de la planta. Este mecanismo es compartido con otros patógenos vasculares que también invaden el xilema de la planta, como la bacteria Xylella fastidiosa, que actualmente amenaza los cultivos de olivo, almendro, viña, y otras especies en el Mediterráneo.
Pocas opciones de solanáceas resistentes
Los tomates comerciales son susceptibles a Ralstonia solanacearum y las pocas variedades resistentes que existen hacen tomates muy pequeños. En regiones en las que R. solanacearum es un problema, se ha optado por injertar variedades comerciales en el pie de la variedad resistente. Precisamente esta es la solución que han implementado los agricultores del estado de Carolina del Norte (EE.UU.), donde la bacteria produce pérdidas en múltiples cultivos desde hace más de un siglo.
En un intento por comprender mejor las características de la planta que determinan la resistencia a R. solanacearum, investigadores de la Universidad Estatal de Carolina del Norte, contactaron el grupo de investigación del CRAG liderado por Valls y Sanchez Coll. La colaboración entre los primeros, acostumbrados a la investigación aplicada en el campo, y los segundos, expertos en investigación básica de laboratorio, permitió descubrir cuáles son los cuellos de botella que los tomates resistentes imponen a la bacteria para evitar su colonización.
Cuatro cuellos de botella para la bacteria
Para seguir el proceso de infección de Ralstonia en tomate en vivo, los investigadores del CRAG y de la Universidad Estatal de Carolina del Norte utilizaron bacterias modificadas luminiscentes y fluorescentes. Con estas bacterias infectaron diferentes variedades de tomate comerciales: unas muy susceptibles (variedad Marmande), unas de resistencia moderada (Shield), y las plantas muy resistentes que se utilizan para hacer los injertos (Hawaii 7996).
Siguiendo la colonización de R. solanacearum en estas tres variedades vieron que la capacidad de invasión de los tejidos de la planta por parte de la bacteria era diferente.
“Las plantas resistentes tienen la capacidad de bloquear la expansión de la bacteria en cuatro puntos diferentes: en la misma raíz, donde la bacteria no logra expandirse, en los vasos conductores que transportan el agua y las sales minerales, en el movimiento horizontal en el haz de vasos del sistema vascular y, finalmente, en el movimiento radial de la bacteria desde los vasos a los tejidos adyacentes.”, explica Marc Planas-Marqués, estudiante de doctorado del CRAG y co-primer autor del artículo.
Es la primera vez que se estudia de manera tan sistemática el proceso de infección, y los investigadores se han sorprendido al descubrir que Ralstonia solanacearum tiene la capacidad también de salir de los vasos e infectar los tejidos cercanos.
“Es un descubrimiento muy relevante, ahora sabemos que cuando la infección está muy avanzada la bacteria sale del vaso para nutrirse de las células de la planta, lo que le puede dar más capacidad de multiplicación y de infección a un nuevo huésped”, explica Marc Valls.
“Este trabajo tiene implicaciones apasionantes para la mejora genética de las solanáceas, ya que ahora tenemos un contexto unificado que describe los campos de batalla entre el patógeno y la planta. Además, esta investigación ha desarrollado nuevas herramientas y técnicas para medir los mecanismos de resistencia de manera directa en la planta entera, lo que permitirá estudios futuros que conecten cada uno de estos mecanismos con la genética que hay detrás”, añade el co-primer autor del trabajo Jonathan Kressin, investigador de la Universidad Estatal de Carolina del Norte que hizo una estancia de investigación en CRAG.
Volviendo al símil de las enfermedades humanas, ahora los investigadores ya saben a qué órganos afecta el patógeno y qué características tienen los huéspedes más resistentes. En este sentido es importante destacar que el trabajo se ha hecho en variedades comerciales. A partir de estos resultados, se deberá devolver al trabajo molecular y genético y seguir muy vigilantes para contener la expansión de este patógeno en nuestros campos.
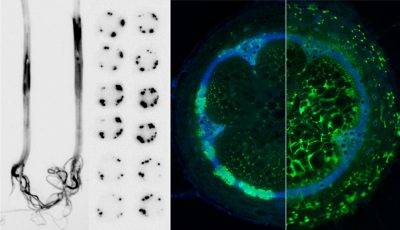
- La invasión de la bacteria en las raíces y los vasos conductores se puede seguir gracias al uso de bacterias luminiscentes (imágenes de la derecha donde la bacteria se visualiza en color negro) y bacterias fluorescentes (imágenes de la izquierda donde la bacteria se ve en color verde) (Crédito: Marc Planas-Marqués, CRAG)
- Tomates infectados con Ralstonia solanacearum. A la izquierda una planta de tomate susceptible a la infección y a la derecha una de naturalmente resistente. (Crédito: Marc Planas-Marqués, CRAG)
Artículo de referencia
Marc Planas-Marquès, Jonathan P Kressin, Anurag Kashyap, Dilip R Panthee, Frank J Louws, Nuria S Coll, Marc Valls. Four bottlenecks restrict colonization and invasion by the pathogen Ralstonia solanacearum in resistant tomato. Journal of Experimental Botany, 71(6), 2157–2171. DOI:10.1093/jxb/erz562
Sobre los autores y la financiación del estudio
El estudio es fruto de una colaboración entre investigadores del Centro de Investigación en Agrigenómica (CRAG, Barcelona, España) y la North Carolina State University (EE.UU.). El trabajo experimental ser ha llevado a cabo en el CRAG y ha sido financiado por fondos de Ministerio de Economía y Competitividad y por la Generalitat de Catalunya y por una beca del International Fellowship-Indian Council of Agricultural Research. Los autores de EE.UU. han sido financiados por fondos del National Institute of Food and Agriculture, de la North Carolina Tomato Growers Association y por la beca Monsanto Graduate Fellowship.
Sobre el Centro de Investigación en Agrigenómica (CRAG)
El Centro de Investigación en Agrigenómica (CRAG) es un centro que forma parte del sistema CERCA de la Generalidad de Cataluña, y que se estableció como consorcio de cuatro
instituciones: el Consejo Superior de Investigaciones Científicas (CSIC), el Instituto de Investigación y Tecnología Agroalimentarias (IRTA), la Universidad Autónoma de Barcelona (UAB) y la Universidad de Barcelona (UB). La investigación del CRAG se extiende desde la investigación básica en biología molecular de plantas y animales de granja, a las aplicaciones de técnicas moleculares para la mejora genética de especies importantes para la agricultura y la producción de alimentos en estrecha colaboración con la industria. En el año 2016, el CRAG obtuvo el reconocimiento de “Centro de Excelencia Severo Ochoa9” por el Ministerio de Economía y Competitividad.
Informaciones relacionadas
Cultivos resistentes a la sequía para asegurar el abastecimiento alimentario
Investigación aporta herramientas para la mejora genética del melón